Desde que se describió que las secuencias codificantes de la mayor parte de los genes eucariotas están interrumpidas por intrones, se hizo patente la necesidad de conocer los mecanismos que controlan su escisión de los transcritos maduros. Diversos trabajos han revelado que el espliceosoma, un complejo ribonucleoproteico constituido por cinco snRNP y más de 300 proteínas auxiliares, es el responsable de este procesamiento a través de un mecanismo denominado splicing. En las últimas décadas, se ha demostrado que este complejo juega un papel esencial en numerosos procesos fisiológicos de los organismos eucariotas, asegurando una correcta expresión génica en respuesta a estímulos tanto internos como externos.
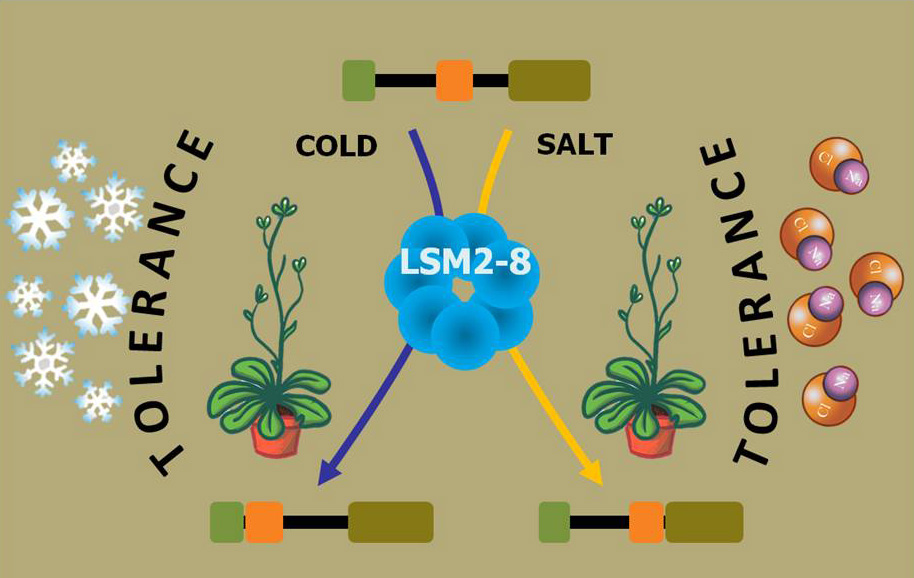
En el trabajo publicado recientemente en Nucleic Acids Research por el grupo del Prof. Julio Salinas, se aporta una nueva perspectiva de los mecanismos que controlan la actividad del espliceosoma mediante estudios realizados con la planta modelo Arabidopsis thaliana. Hasta el día de hoy, se asumía que la actividad y especificidad del espliceosoma estaba modulada por la acción de sus proteínas auxiliares. En este estudio, se presentan resultados que rompen con este paradigma, demostrando que un componente estructural del espliceosoma, como es el complejo LSM2-8 del snRNP U6, determina la especificidad de su actividad. Más aún, se presentan evidencias de que este complejo es esencial para el control del splicing, tanto constitutivo como alternativo, de transcritos específicos en función de las condiciones ambientales y para la correcta adaptación de las plantas a su entorno.
Todos estos resultados han puesto de manifiesto una función inesperada para el complejo LSM2-8, que representa un nuevo nivel de regulación posttranscripcional de la respuesta a estímulos externos en organismos eucariotas.
Referencia: Environment-dependent regulation of spliceosome activity by the LSM2-8 complex in Arabidopsis. Cristian Carrasco-Lopez, Tamara Hernandez-Verdeja, Carlos Perea-Resa, David Abia, Rafael Catala and Julio Salinas. Nucleic Acids Research, 2017. doi: 10.1093/nar/gkx375

