Un artículo publicado en la revista International Journal of Biological Macromolecules por el grupo de RMN Biomolecular del Centro de Investigaciones Biológicas Margarita Salas (CSIC), liderado por el Dr. Francisco J. Blanco, describe la estructura de la proteína ING3 y su interacción con la histona H3. El trabajo contribuye a esclarecer los mecanismos moleculares que podrían explicar los posibles efectos deletéreos de las mutaciones de ING3 detectadas en tumores.
El genoma, el soporte de la información para la vida, se encuentra en el núcleo celular unido a diversas proteínas formando la cromatina. La mayoría de estas proteínas son histonas, que organizan el ADN de forma más o menos compacta. El grado compactación (y, por tanto, su accesibilidad) depende de modificaciones en las histonas. Las proteínas de la familia ING (INhibitors of Growth) se unen a la cromatina y reconocen específicamente la metilación de la histona H3 en la posición K4. De esta forma reclutan enzimas que producen modificaciones adicionales (acetilación o desacetilación) en las histonas. El conjunto de modificaciones de las histonas (así como del propio ADN) determina en qué medida se expresan los genes, y un patrón de modificaciones anómalo puede causar patologías como el cáncer.
ING3 es la menos estudiada de las cinco proteínas ING humanas. Aunque es necesaria para el desarrollo embrionario, su exceso estimula la proliferación celular y se correlaciona con un mal pronóstico en pacientes con cáncer de próstata.
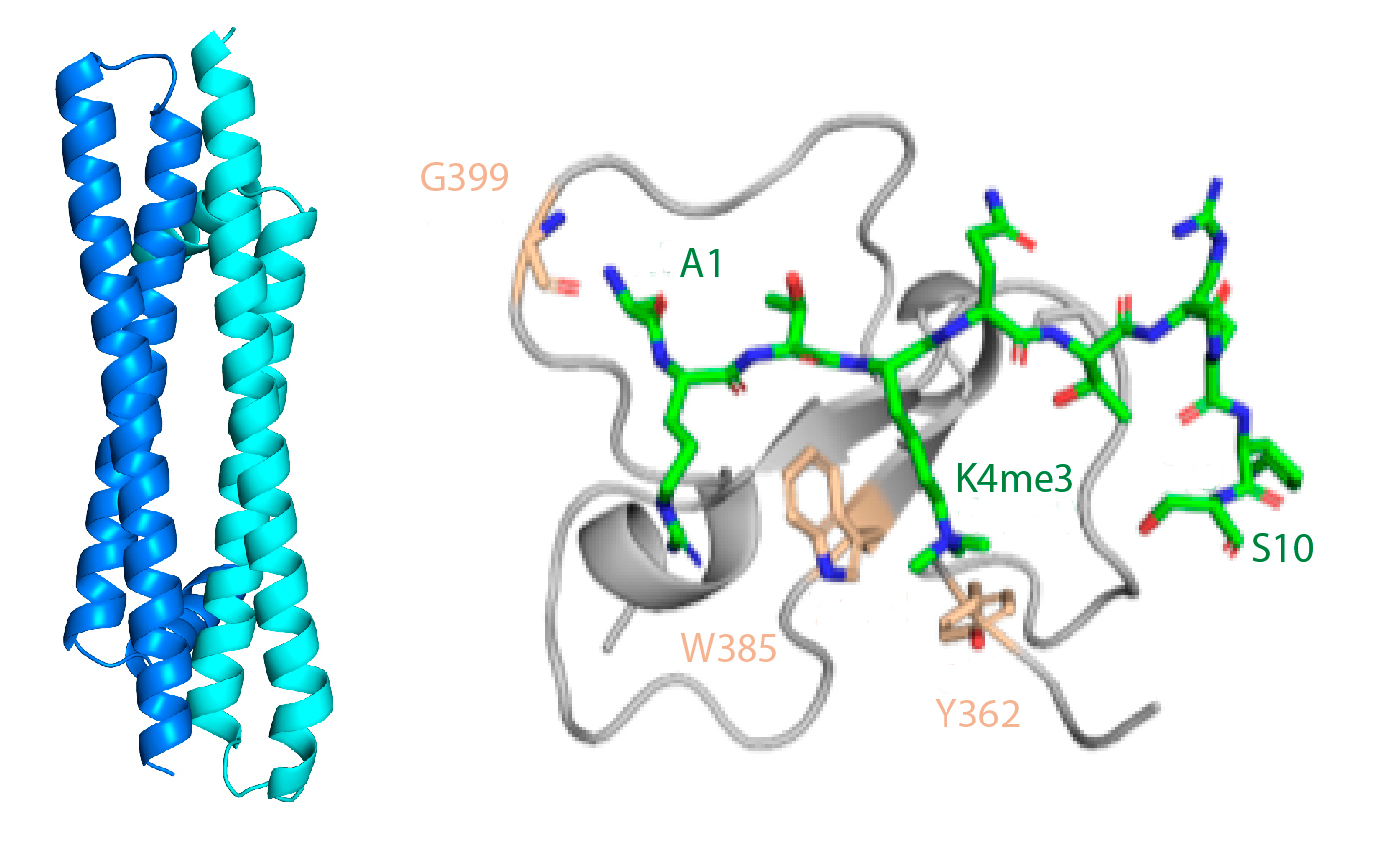
Mediante RMN y cristalografía, Ferreras-Gutiérrez et al. han encontrado que dos moléculas de ING3 se ensamblan en una estructura de hélices superenrolladas con una orientación antiparalela. Una larga región central desordenada separa la región helicoidal de la que se une a la histona, por lo que ING3 es un lector bivalente de la metilación en histona H3, incrementándose así la afinidad de la interacción. Esta estructura explica por qué ciertas mutaciones en ING3 aparecen con alta frecuencia relativa en tumores. Algunas de ellas desestabilizan su estructura y otras interfieren con el reconocimiento de la histona.
La organización estructural de ING3 es muy similar a la de sus homólogos ING4 e ING5. En el caso de ING1 e ING2 aún no se conoce en detalle su estructura. Aunque ING1 e ING2 son supresores de tumores, ING3 es una oncoproteína, mientras que ING4 e ING5 no se pueden clasificar con claridad en una de estas dos categorías. Serán necesarios nuevos estudios estructurales y funcionales para aclarar estos y otros aspectos desconocidos de la familia de proteínas ING.
Referencia: Structural analysis of ING3 protein and histone H3 binding. Mariola Ferreras-Gutiérrez, Belén Chaves-Arquero, Amaia González-Magaña, Nekane Merino, Ignacio Amusategui-Mateu, Sonia Huecas, Francisco J. Medrano, Francisco J. Blanco (2023) International Journal of Biological Macromolecules, Vol 242, Part 1, 124724. https://doi.org/10.1016/j.ijbiomac.2023.124724.

