Un trabajo publicado en la revista Journal of Medical Virology por el grupo liderado por María Montoya en el Centro de investigaciones Biológicas Margarita Salas (CSIC) demuestra el impacto de proteínas accesorias del virus SARS-CoV-2 sobre el metabolismo y la función mitocondrial de células epiteliales de pulmón. El estudio describe el papel que juegan cuatro de estas proteínas, ORF3a, ORF9b, ORF9c y ORF10, en la reprogramación metabólica de estas células pulmonares y muestra el potencial del modelo bioinformático GSMM para investigar la patogénesis del virus causante de la COVID-19.
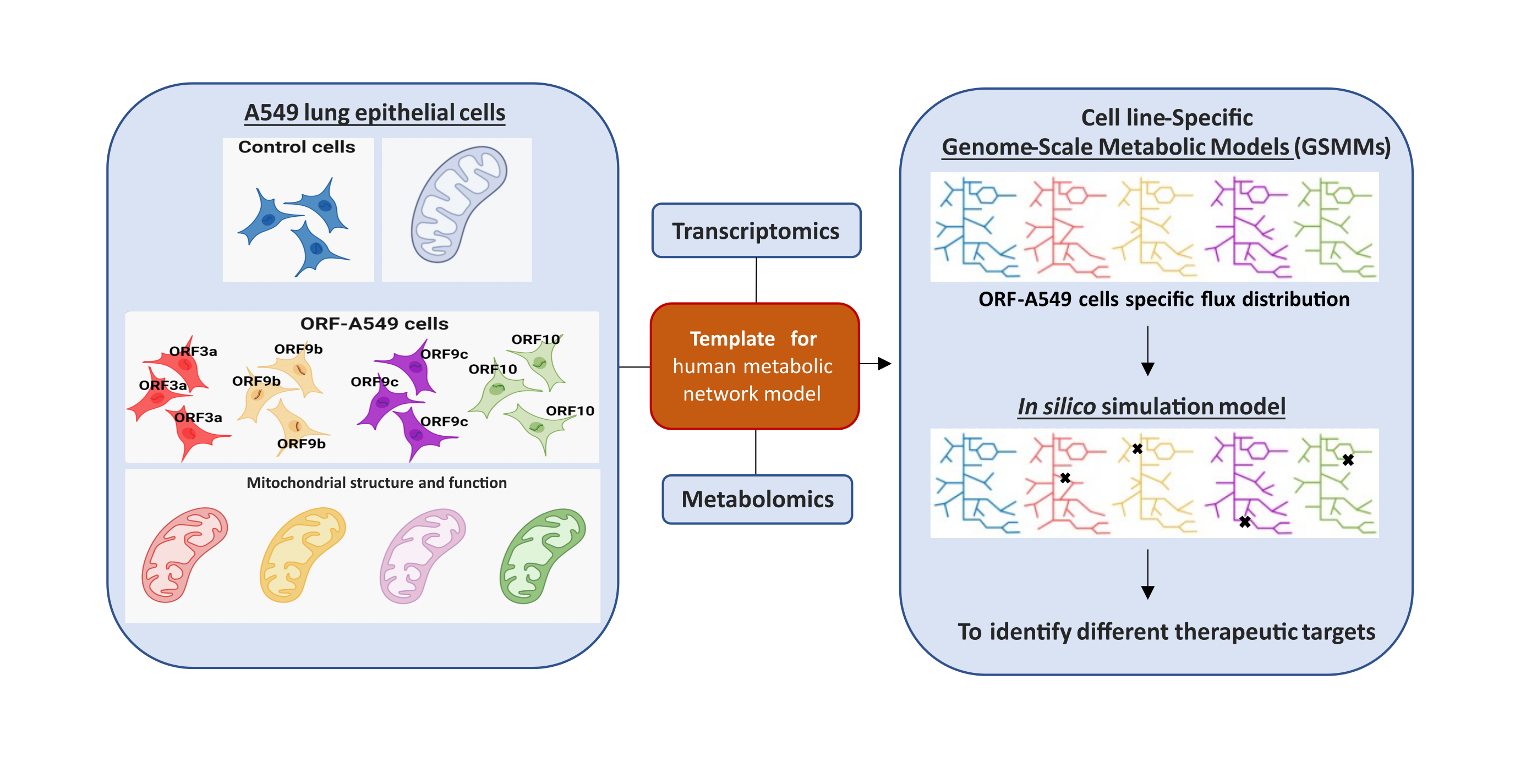
Entre las proteínas menos conocidas del virus SARS-CoV-2 se encuentran 11 proteínas denominadas accesorias que, aunque no tienen una relación directa con la replicación viral y la formación de nuevas partículas virales, si parecen involucradas en las estrategias de evasión del sistema inmune por parte del virus, en las interacciones con rutas metabólicas y en la inducción de la muerte celular. De entre ellas, los investigadores seleccionaron ORF3a, ORF9b, ORF9c y ORF10 para estudiar su efecto sobre células epiteliales de origen pulmonar (A549).
López-Ayllón et al. generaron líneas celulares transducidas con cada una de estas proteínas para estudiar la respuesta celular inducida por ellas de forma independiente mediante análisis de transcriptómica y metabolómica, complementados con diversos ensayos funcionales. Además, se utilizó un modelo bioinformático conocido como Modelo Metabólico a Escala Genómica (GSMM, por sus siglas en inglés, Genome-Scale Metabolic Model). La aplicación de este modelo para estudiar el impacto de las proteínas accesorias en el metabolismo celular de la célula huésped, es una aportación innovadora de este estudio, ya que previamente solo se había llevado a cabo con el virus completo.
Los resultados pusieron de manifiesto la alteración de los flujos metabólicos en rutas relacionadas con el metabolismo de lípidos, de glúcidos, de proteínas y de nucleótidos. La expresión de ORF3a alteró mayoritariamente rutas del metabolismo lipídico, mientras que ORF9b, ORF9c y ORF10, tuvieron un mayor efecto en el metabolismo de los aminoácidos.
Modelos de simulación in silico para analizar la modulación de determinadas moléculas o metabolitos que pudieran revertir la alteración metabólica observada, permitieron identificar diferentes dianas terapéuticas. De entre las dianas encontradas para las células transducidas con ORF3a, se validó el modelo con un inhibidor de la fosfolipasa D2 (PLD2). Tras el tratamiento de las células con este inhibidor, se pudo recuperar el fenotipo mitocondrial y metabólico de las mismas, asemejándose al observado en células control.
La metodología presentada en este trabajo permite destacar la utilidad de los GSMM para el análisis de células transducidas únicamente con una proteína viral, que podría convertirse en una herramienta válida para investigar tanto la patogénesis del virus SARS-CoV-2 como para la búsqueda de nuevas estrategias antivirales.
Referencia: Metabolic and mitochondria alterations induced by SARS-CoV-2 accessory proteins ORF3a, ORF9b, ORF9c and ORF10. Blanca D. López-Ayllón, Silvia Marin, Marco Fariñas Fernández, Tránsito García-García, Raúl Fernández-Rodríguez, Ana de Lucas-Rius, Natalia Redondo, Laura Mendoza-García, Carles Foguet, Juozas Grigas, Alba Calvet, José Manuel Villalba, María Josefa Rodríguez Gómez, Diego Megías, Biagio Mandracchia, Daniel Luque, Juan José Lozano, Cristina Calvo, Unai Merino Herrán, Timothy M. Thomson, Juan J. Garrido, Marta Cascante and María Montoya (2024) Journal of Medical Virology, 96:e29752. DOI:10.1002/jmv.29752

