Un trabajo publicado en Cell Mol. Life Sci. por el equipo liderado por la Dra. Susana Camarero del Grupo de Biotecnología para la Biomasa Lignocelulósica del Centro de Investigaciones Biológicas Margarita Salas (CSIC), ha descrito la optimización de la secuencia líder del factor α para mejorar la producción recombinante de enzimas fúngicas en Saccharomyces cerevisiae.
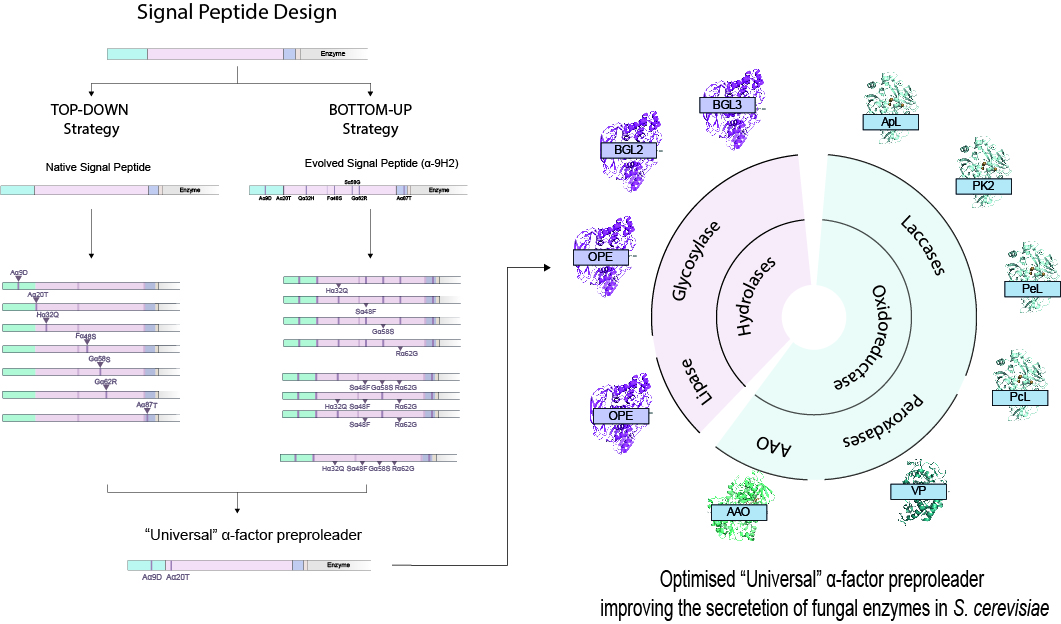
La levadura S. cerevisiae se ha utilizado históricamente como sistema de expresión para proteínas eucariotas debido a su fácil manipulación, bajos requerimientos y capacidad para llevar a cabo modificaciones postraduccionales de las proteínas. Una de las mejores estrategias para potenciar la secreción de proteínas recombinantes es el uso de péptidos señal que determinan la vía de secreción. Entre ellos, la secuencia pre-prolíder de la feromona factor α de S. cerevisiae ha jugado un papel importante en la producción de proteínas recombinantes en levadura. En estudios previos del grupo, el uso de esta secuencia señal en combinación con la evolución dirigida de enzimas ha permitido conseguir la difícil expresión de ciertas lacasas fúngicas en S. cerevisiae, obteniendo diferentes secuencias señales evolucionadas que mejoran la secreción de estas enzimas. Sin embargo, el diseño de un péptido señal universal que incremente la producción de diversas enzimas heterólogas en S. cerevisiae es un desafío pendiente.
En este estudio, los investigadores utilizaron dos estrategias paralelas de ingeniería del pre-prolíder del factor α para mejorar la producción de enzimas recombinantes en S. cerevisiae, analizando el efecto de las mutaciones acumuladas en dicha secuencia a lo largo de sucesivas rondas de evolución dirigida, así como de otras mutaciones conocidas, y sus posibles interacciones epistáticas. Ambos enfoques coincidieron en el efecto sinérgico positivo de cuatro mutaciones contenidas en la secuencia líder optimizada final, que mejoraron notablemente la secreción de varias oxidorreductasas e hidrolasas fúngicas. Además, los autores proporcionan unas directrices adicionales para mejorar aún más la producción heteróloga de una enzima particular, mediante la mutagénesis saturada combinatorial de dos posiciones concretas de la secuencia líder optimizada fusionada a la proteína de interés.
Alcanzar niveles suficientes de producción de enzimas recombinantes en sistemas de expresión heterólogos es un paso necesario para llevar a cabo experimentos de ingeniería de proteínas, además de ser un requisito indispensable para cualquier ensayo de aplicación o la comercialización del producto. Teniendo en cuenta que S. cerevisiae es uno de los sistemas de expresión de enzimas fúngicas más utilizados, los resultados obtenidos en este estudio ofrecen un importante avance para mejorar la secreción de proteínas, proporcionando un péptido señal optimizado de carácter universal para ser utilizado en este contexto.
Este trabajo se ha realizado en el marco del Proyecto Europeo WoodZymes, coordinado por la Dra Susana Camarero, donde la función del grupo de Biotecnología para la Biomasa Lignocelulósica es la evolución dirigida de lacasas fúngicas para obtener biocatalizadores con propiedades extremófilas, capaces de trabajar en las condiciones altamente exigentes de pH y temperatura elevada requeridas por la industria de transformación de la madera. Los resultados obtenidos contribuirán a incrementar los niveles de producción de los biocatalizadores diseñados. El proyecto WoodZymes ha recibido financiación de Bio Based Industries Joint Undertaking, que forma parte del programa de investigación e innovación Horizonte 2020 de la Unión Europea, en virtud del acuerdo de subvención número 792070.
Referencia: Design of an improved universal signal peptide based on the α-factor mating secretion signal for enzyme production in yeast. Aza P, Molpeceres G, de Salas F and Camarero S (2021) Cell Mol. Life Sci. https://doi.org/10.1007/s00018-021-03793-y

